유전 요인과 생활환경 요인의 상호작용이 제2형 당뇨병 발생에 미치는 영향: 한국인유전체역학 조사사업(KoGES) 자료를 이용하여
Effects of gene-lifestyle environment interactions on type 2 diabetes mellitus development: an analysis using the Korean Genome and Epidemiology Study data
Article information
Trans Abstract
Purpose
This study focused on identifying the interaction effects of genetic and lifestyle-environmental factors on the development of type 2 diabetes mellitus (T2D).
Methods
Study subjects were selected from the Korean Genome and Epidemiology Study (KoGES) from 2001 to 2014. Data on genetic variations, anthropometric measurements, biochemical data, and seven lifestyle factors (diet, physical activity, alcohol drinking, smoking, sleep, depression, and stress) were obtained from 4,836 Koreans aged between 40 and 59 years, including those with T2D at baseline (n = 1,209), newly developed T2D (n = 1,298) and verified controls (n = 3,538). The genetic risk score (GRS) was calculated by using 11 single-nucleotide polymorphisms (SNPs) related to T2D development and the second quartile was used as the reference category. A Cox proportional hazards regression model was used to evaluate the associations of GRS and lifestyle factors with T2D risk, controlling for covariates.
Results
Multivariate regression analysis revealed that GRS was the strongest risk factor for T2D, and body mass index (BMI), smoking, drinking, and spicy food preference also increased the risk. Lifestyle/environmental factors that showed significant interactions with GRS were BMI, current smoking, current drinking, fatty food preference, and spicy food preference.
Conclusions
Interactions between genetic factors and lifestyle/environmental factors were associated with an increased risk of T2D. The results will be useful to provide a new perspective on genetic profiling for the earlier detection of T2D risk and clues for personalized interventions, which might be more effective prevention strategies or therapies in individuals with a genetic predisposition to T2D.
서론
당뇨병 환자는 전세계적으로 지속적인 증가 추세에 있으며 2019년 20-79세 성인 당뇨병 환자의 유병률은 9.3%이었고 2023년에는 그 수가 5억 7천 8백만명에 이를 것으로 추산되고 있다[1]. 당뇨병의 90% 이상을 차지하는 제2형 당뇨병은 인슐린 저항성이나 췌장 베타세포의 인슐린 분비 장애에 의해서 발생하며, 유전적 요인과 환경적 요인의 상호작용으로 발생하는 대사질환으로 알려져 있다[2]. 최근 제2형 당뇨병에서 인슐린 결핍이나 인슐린 저항성을 유발하며 당뇨병의 발생에 감수성을 나타내는 유전자들이 보고되고 있는데[3], 유전자의 변이는 인종에 따라 다른 결과가 나타날 가능성이 높아[4] 특정 인구에 따른 유전적 요인을 찾는 노력이 이루어지고 있다. 이에 Go 등[5]은 한국인을 대상으로 한 대규모 코호트 자료를 이용하여 제2형 당뇨병과 관련된 19개의 단일염기다형성(single nucleotide polymorphism, SNPs)을 보고하였다.
제2형 당뇨병 발생에 영향을 미치는 환경적 요인은 주로 생활습관과 관련된 요인으로 에너지 섭취, 섭취영양성분, 식습관, 신체활동, 알코올 섭취, 흡연, 수면, 우울, 스트레스 등이 알려져 있다[6,7]. 특히 에너지 섭취의 증가와 에너지 소비의 감소는 비만으로 이어지는데 이러한 비만한 상태는 인슐린 저항성으로 진행되는 경우가 많아 T2D발생의 주요한 위험요인이 된다[1]. 제2형 당뇨병은 유전적 요인과 생활환경 요인의 상호작용으로 발생률이 상승될 수 있어[8], 유전과 생활환경의 상호작용을 파악하는 것은 유전적 위험요인이 있는 대상자들의 질병발생 위험도를 예측하고 위험한 생활환경의 변화를 통한 질병예방에 활용될 수 있다[9].
그러나 제2형 당뇨병 발생에 있어서 유전과 환경의 상호작용을 조사한 몇몇 선행연구는 환경요인으로 특정 영양성분이나 활동량만 포함하여 생활환경 요인을 포괄적으로 파악하지 못하였고[10,11], 환경요인과 한 두가지의 SNPs과의 상호작용만을 분석하여 제2형 당뇨병 발생과 관여하는 여러 SNPs의 복합적인 상호작용을 조사하지 못하였다[12]. 무엇보다 이러한 유전적 요인과 생활환경 요인은 인종과 지역에 따라 차이가 있으므로 한국인에게 적합한 제2형 당뇨병 예방중재의 개발을 위해서는 한국인을 대상으로 한 연구가 필수적이나 국내 선행연구는 활동량의 감소나 섭취영양소 등 일부 생활환경 요인을 제2형 당뇨병 발생의 위험요인으로 보고한 정도이다[13].
따라서 본 연구에서는 한국인유전체역학조사사업(Korean Genome and Epidemiology Study [KoGES]) 지역사회기반 코호트 자료(2001년∼2014년)를 이용하여 제2형 당뇨병 발생에 영향을 미치는 것으로 보고된 SNPs을 검증하고 이를 활용하여 유전적 위험도 점수(genetic risk scores, GRS)를 산출하여 GRS와 다양한 생활환경 요인의 상호작용이 제2형 당뇨병 발생에 미치는 영향을 파악하고자 한다.
연구 방법
1. 연구설계
본 연구는 한국인유전체역학조사사업에서 실시한 전향적 지역사회기반 코호트 자료를 이용하여 제2형 당뇨병 발생의 유전적 요인과 생활환경 요인의 상호작용을 파악하기 위한 서술적 상관관계 연구이다.
2. 연구대상
본 연구는 2001년부터 2014년까지 2년 단위로 기반조사(1기) 및 6차 추적조사(7기)를 실시한 질병관리본부의 KoGES에서 진행된 지역사회기반 코호트자료를 사용하였다. KoGES는 40∼69세 남·여 일반 인구집단을 대상자로 하여, 사업 참여와 시료 수집에 대한 동의를 얻은 후 건강이나 질병력, 운동, 식습관 등 생활습관과 관련한 다양한 설문조사와 신체계측 및 검진을 통해 자료를 수집하고, 혈액과 소변을 채취하여 임상검사 자료와 인체자원(혈청, 혈장, DNA)을 확보하여, 2∼4년 주기로 반복 추적하는 코호트 사업이다[14].
World Health Organization (WHO)에서 제시한 제2형 당뇨병 진단기준에 따른 제2형 당뇨병 대상자 선정기준은 다음과 같다. 1) 공복혈당 ≥ 126mg/dl; 2) 75g 경구 당 부하검사에서 2시간 후 혈당 ≥ 200mg/dl; 3) HbAIc ≥ 6.5%; 4) 질병발병 시점 ≥ 40세. 대조군의 선정기준은 다음과 같다. 1) 공복혈당 <100mg/dl; 2) 5g 경구 당 부하검사에서 2시간 후 혈당 < 140mg/dl; 3) HbAIc < 5.7%. 4) 제2형 당뇨병의 질병력이 없음.
본 연구의 대상자 선정과정은 Figure 1과 같다. 기반조사의 전체 응답자는 10,031명이었으며, 이 중 유전자 정보가 없는 1,190명, 혈액검사 및 당뇨병 질병력이 누락된 273명은 제외하였으며 분석에 사용된 대상자는 총 8,568명이었다. 대조군은 기반조사 8,568명 중 이미 제2형 당뇨병이 발생한 1,209명과 추적과정에서 제2형 당뇨병이 발생한 1,298명을 제외하고, 대상자 선정기준에서 대조군의 기준에 부합하지 않는 2,523명을 제외하여 최종 3,538명이었다. 추적 제2형 당뇨병 발생군은 기반자료에서 당뇨병이 발생한 1,209명을 제외하고 1차∼6차까지 추적과정에서 제2형 당뇨병이 발생된 대상자들로 총 1,298명이었다. 제2형 당뇨병군은 기반자료에서 제2형 당뇨병이 이미 발생한 대상자 1,209명과 추적 제2형 당뇨병 발생군 1,298명을 모두 포함하여 총 2,507명이었다. 유전적 특성은 변하지 않는 개인의 고유한 특성이므로 기반자료에서 제2형 당뇨병이 이미 발생한 대상자들도 제2형 당뇨병군에 포함하였으나, 제2형 당뇨병 발생에 대한 유전과 생활환경의 상호작용을 파악하기 위해서는 기반자료에서 이미 당뇨병이 발생한 대상자들의 환경적인 영향을 확인할 수 없으므로 추적 제2형 당뇨병 발생군만 포함하였다.
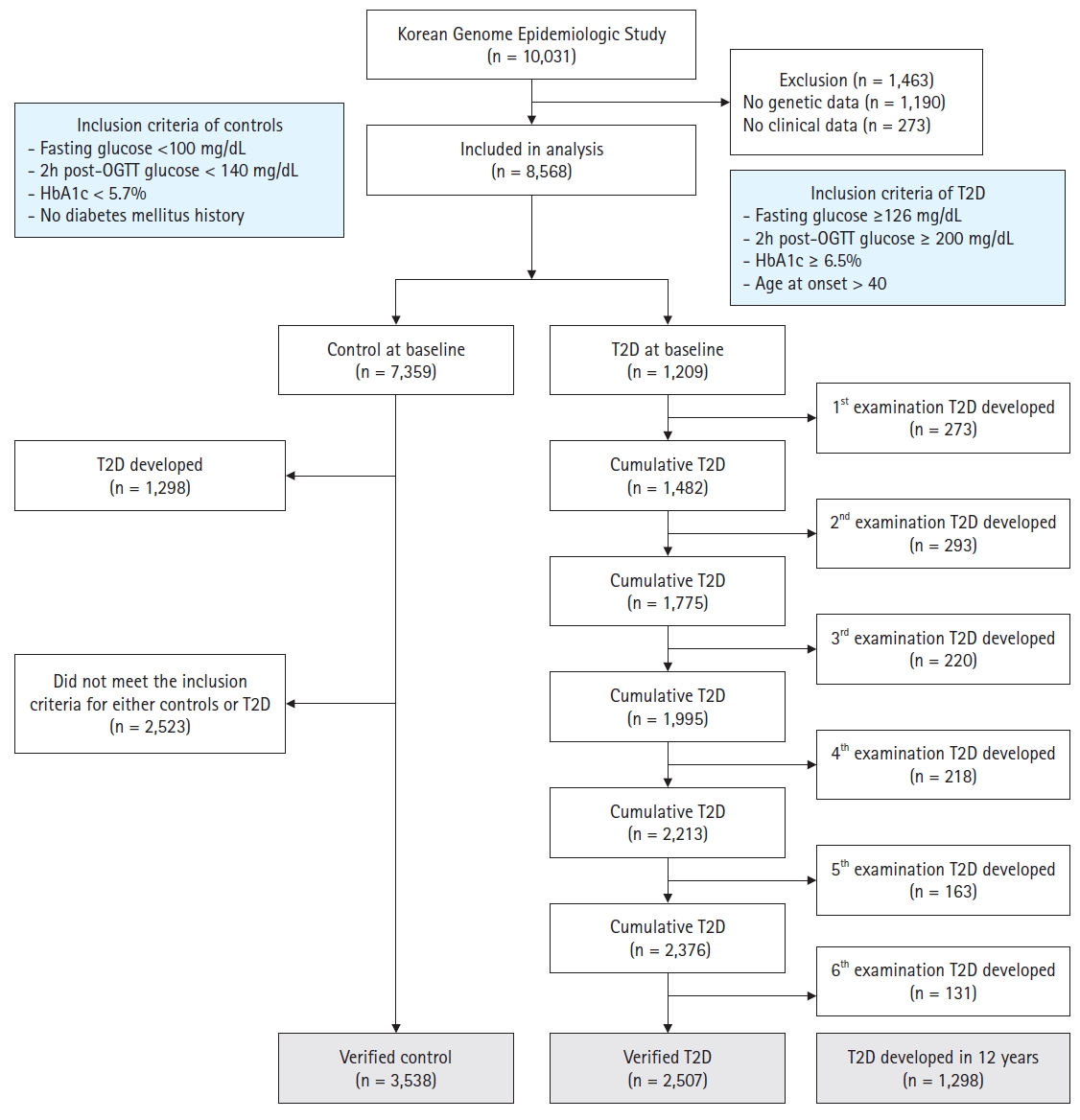
Overview of subjects for this study. HbA1c=hemoglobin A1c; OGTT=oral glucose tolerance test; T2D=type 2 diabetes mellitus.
본 연구에 사용되는 데이터가 유전과 생활환경의 상호작용 분석에 충분한 표본[15]인지 확인하기 위하여 검정력 통계프로그램 QUANTO ver. 1.2.4를 이용하여 표본수의 적정성을 평가하였다. 유의수준(α) .05, 검정력(β) .80, 위험대립유전자 빈도 0.70, 환경요인 노출확률 0.45, 질병 발생률 0.10로 가정하였으며[16], 그 결과 필요한 최소 표본 수는 2,183명으로 본 연구의 상호작용 분석에 사용된 총 대상자의 수 4,836명은 적정성을 충족하는 것으로 나타났다.
3. 윤리적 고려
본 연구는 질병관리본부 국립중앙인체자원은행(승인번호: 2018-042)과 계명대학교 연구윤리심의를 거쳐(40525-201802 -HR-116-02) 진행되었다.
4. 생활환경 요인(Lifestyle-environmental factors)
에너지 섭취량(Kcal)과 섭취영양성분을 확인하기 위하여 Food Frequency Questionnaire (FFQ)를 이용하였으며 조사일로부터 1년 전까지의 기간 동안 제시된 총 106개 식품의 섭취빈도와 섭취량을 조사하고. 섭취빈도와 섭취량을 이용하여 1일 평균 에너지, 단백질, 지방, 탄수화물, 칼슘, 나트륨, 콜레스테롤을 산출하였다[17]. 식사 선호도에 대한 문항은 총 6개로 식사의 규칙성, 과식빈도, 식사속도, 짠 음식 선호도, 매운 음식 선호도, 기름진 음식 선호도로 구성되었다.
에너지 소비량(Metabolic equivalent of task, MET)은 하루 일과 중 육체적 활동에 따른 시간을 에너지 소비량을 나타내는 점수를 나타낸 것이며[18], MET = ∑[신체활동 강도에 대한 대사당량 값 × 소요시간]으로 계산된다.
음주에 대한 문항은 음주여부 및 음주기간, 음주량을 포함하며, 음주여부는 현재음주, 과거음주, 음주경험 없음으로 구분된다. 음주량은 한 번 술을 마실 때의 평균 섭취량을 술의 종류와 섭취량(잔)으로 계산하고 총 섭취 알코올량(g)으로 환산한 수치이다. 흡연에 대한 문항은 흡연여부 및 흡연기간, 총 흡연량을 포함하며, 흡연여부는 현재흡연, 과거흡연, 흡연경험 없음으로 구분된다. 총 흡연량은 갑년단위로 환산하였으며, 하루에 한 갑 담배를 피웠다고 가정했을 때 총 흡연한 년 수를 의미한다.
수면은 평소 수면 시간과 불면증 여부에 대한 질문으로 구성되었으며 불면증은 ‘예’, ‘아니오’로 응답하며, 평소 수면 시간은 지난 1년간 하루 평균 수면 시간에 대한 값이며 시간단위로 조사되었다. 우울은 Beck Depression Inventory [19]로 측정되었으며, 스트레스는 사회심리적 스트레스 척도의 단축형 Psychological Wellbeing Index – Short Form [20]으로 측정되었다.
5. 유전적 요인
유전 데이터는 KoGES의 유전체역학과에서 Affymetrix Genome-Wide Human SNP array V.5.0 (Affymatrix, Santa Clara, California, USA)를 이용하여 판별하고 질관리가 된 자료를 이용하였다[21]. 본 연구에서는 한국인에서 제2형 당뇨와 유의한 상관성이 있는 것으로 보고된 18개의 SNPs을 이용하여 분석하였고[5]. 유전적 위험도 점수는 본 연구에서 제2형 당뇨병과 연관성이 있는 것으로 나타난 11개의 SNPs을 대상으로, 각각 비위험 대립 유전자가 두 개 있는 경우 0점, 이형 대립유전자가 있는 경우 1점, 위험 대립 유전자만 두 개 있는 경우 2점으로 하여 총 11개 SNPs의 점수를 합산하여 계산하였다. 이는 선행연구[5]에 따라 각 유전형 빈도를 조우성 모드(co-dominant mode)를 기본으로 재구성한 것으로 교차비 분석을 위하여 제2형 당뇨병군에서 GRS의 중분위수를 계산하였으며 11점 이상과 10점 이하로 구분하였다.
6. 자료분석
본 연구에서 수집된 자료는 SPSS 22.0 프로그램(IBM Corp., Armonk, NY, USA)을 이용하여 다음과 같이 분석하였다.
1) 추적 제2형 당뇨병 발생군과 대조군의 일반적 특성의 차이를 확인하기 위하여 chi-square test와 independent t-test로 분석하였다.
2) 제2형 당뇨병군과 대조군의 유전적 요인을 확인하기 위하여 18개의 SNPs의 유전자형에 따라 점수를 부여하여 로지스틱 회귀분석을 실시하였으며 제2형 당뇨병 발생에 대한 교차비와 95% confidence interval (CI)로 분석하였다. 본 연구에서는 선행문헌에 근거하여 GRS의 통계적 효과를 극대화하기 위해서 유의수준 <.05를 가지는 모든 SNPs을 분석에 포함시켰다.
3) 제2형 당뇨병군과 대조군의 유전형의 차이와 GRS의 차이는 chi-square test와 independent t-test로 분석하였다.
4) 제2형 당뇨병 발생에 대하여 GRS와 생활환경 요인이 미치는 영향을 확인하기 위하여 Cox 비례위험모형을 사용하여 단변량과 다변량 분석을 시행하였다. 다변량 분석에서 제2형 당뇨병 발생에 대한 위험비(hazards ratio)가 시간의 흐름에 관계없이 일정하다는 비례위험 가정을 평가하여 분석의 타당성을 확보하기 위해 log-minus-log survival plot을 사용하였다.
5) 제2형 당뇨병 발생에서 GRS와 생활환경 요인 사이에 상호작용을 확인하기 위하여 로지스틱 회귀분석을 이용하여 유전과 환경의 상호작용의 교차비로 분석하였다.
6) 유전자형 분포는 Hardy-Weinberg equilibrium (HWE) test따라 분석하였다.
연구 결과
1. 추적 제2형 당뇨병 발생군과 대조군의 일반적 특성
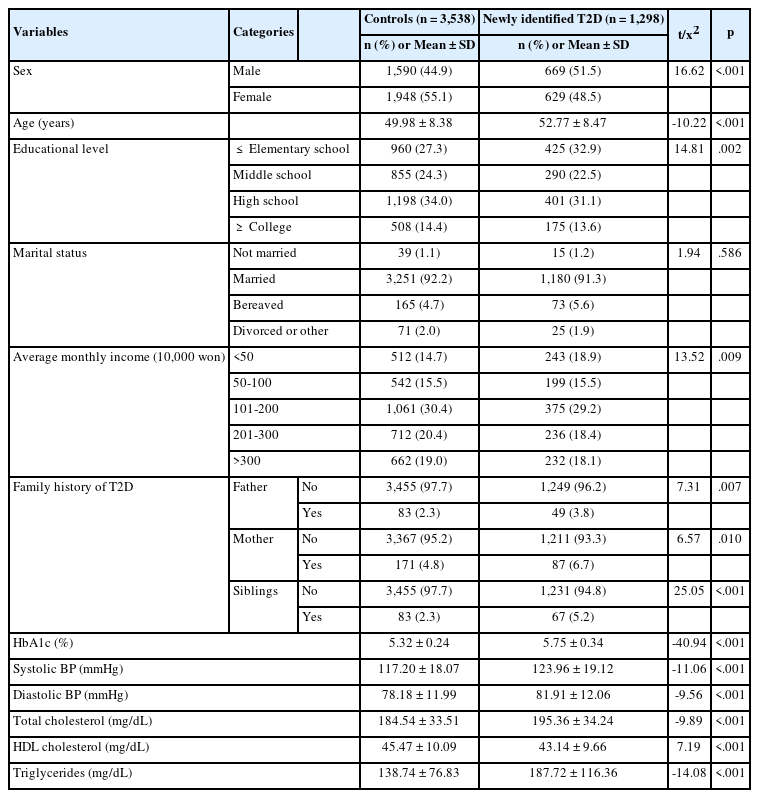
추적 제2형 당뇨병 발생군과 대조군은 성별(p < .001), 나이(p < .001), 교육수준(p = .002)과 월평균수입(p = .009)에서 유의한 차이가 있었다(Table 1). 제2형 당뇨병 가족력은 추적 제2형 당뇨병 발생군이 대조군에 비해 부(p = .007), 모(p = .010), 형제(p < .001)의 가족력이 더 높았다. 당화혈색소(p < .001), 수축기 혈압(p < .001)과 이완기 혈압(p < .001). 총콜레스테롤(p < .001), 중성지방(p < .001)은 추적 제2형 당뇨병 발생군이 대조군보다 더 높았으나, 고밀도 콜레스테롤은 대조군이 더 높았다(p < .001).
2. 제2형 당뇨병과 관련된 18개의 SNPs의 교차비 및 GRS
제2형 당뇨병이군 2,507명과 대조군 3,538명에서 18개 SNPs의 유전자형에 따른 교차비를 확인한 결과 제2형 당뇨병 발생에 위험도가 유의하게 나타난 SNPs은 모두 11개이며, 2개의 SNPs (rs10811661, rs11257655)은 대조군에서 HWE 상태에 위배되는 것으로 나타났다(Supplementary Table 1). 제2형 당뇨병군과 대조군 사이에 유의한 차이를 보인 11개의 SNPs을 이용하여 계산한 GRS는 대조군이 10.22 ± 2.17, 제2형 당뇨병군이 10.83 ± 2.31로 제2형 당뇨병군에서 유의하게 높았다(p < .001) (Supplementary Table 1).
3. 추적 제2형 당뇨병 발생군과 대조군의 생활환경 요인의 차이
추적 제2형 당뇨병 발생군은 식사습관에서 짠 음식 선호도(p =. 001)와 매운 음식 선호도(p < .001), 기름진 음식 선호도(p = .010)에서 대조군보다 높은 선호도를 보였다(Table 2). 대사당량과 현재 운동여부에 있어 두 군 간의 차이는 없었으나 체질량지수는 추적 제2형 당뇨병 발생군이 유의하게 높았다(p < .001). 추적 제2형 당뇨병 발생군과 대조군은 음주여부(p = .017)에서 차이가 있었으며 음주량에서는 추적 제2형 당뇨병 발생군이 유의하게 음주량이 많았다(p = .002). 흡연 여부도 추적 제2형 당뇨병 발생군과 대조군의 차이가 유의하였으며(p < .001), 총 흡연량에서는 추적 제2형 당뇨병 발생군이 유의하게 높게 나타났다(p < .001). 불면증은 두 군 간에 유의한 차이가 있었으나(p = .048), 수면시간에서는 차이가 없었고, 우울은 추적 제2형 당뇨병 발생군이 더 높은 것으로 나타났다(p = .010).
4. 제2형 당뇨병 발생에 영향을 미치는 요인
제2형 당뇨병 발생에 영향을 미치는 유전요인과 생활환경 요인을 확인하기 위해 단변량 Cox 비례위험모형 분석을 한 결과 GRS가 높아질수록 1.40배(95% CI: 1.25-1.56), 짠 음식을 선호할수록 1.14배(95% CI: 1.02-1.27), 매운 음식을 선호할수록 1.19배(95% CI: 1.06-1.33), 기름진 음식을 선호할수록 1.17배(95% CI: 1.02-1.34), BMI가 높아질수록 1.12배(95% CI: 1.10-1.14), 비흡연에 비해 현재흡연은 1.26배(95% CI: 1.05-1.52), 과거흡연은 1.38배(95% CI: 1.13-1.69) 제2형 당뇨병 발생에 위험도가 높아지는 것으로 나타났다(Table 3).
다변량 분석은 단변량 분석에서 위험도가 유의한 것으로 나타난 변수들을 투입하여 전진 선택법을 이용하여 시행하였다. 제2형 당뇨병 발생에 영향을 주는 변수는 BMI (p < .001), GRS (p < .001), 현재흡연(p = .003), 과거흡연(p = .012), 매운 음식 선호도(p = .036) 순서로 모형에 추가되었다. BMI가 1kg/m2 증가할수록 제2형 당뇨병의 위험이 1.12배(95% CI: 1.10-1.14) 높아지며, GRS는 1점 증가할수록 1.09배(95% CI: 1.07-1.12) 증가하였다. 비흡연에 비해 과거흡연은 1.30배(95% CI: 1.06-1.58), 현재흡연은 1.32배(95% CI: 1.10-1.59) 제2형 당뇨병 발생 위험이 높아졌다. 매운 음식 선호도는 선호하는 경우가 선호하지 않는 경우에 비해 제2형 당뇨병 발생 위험이 1.13배(95% CI: 1.01-1.26) 증가하였다.
5. 제2형 당뇨병 발생의 유전적 요인과 생활환경 요인의 상호작용
성별과 나이를 통제한 후 제2형 당뇨병 발생에 대한 GRS와 생활환경 요인의 상호작용을 분석한 결과 GRS와 상호작용이 있는 생활환경 요인으로 BMI, 흡연, 음주, 매운 음식 선호도, 기름진 음식 선호도로 나타났다(Table 4). BMI는 GRS와 상호작용의 교차비가 가장 높게 나타난 변수로 BMI가 23미만이면서 GRS가 낮은 경우에 비해 BMI가 25이상이면서 GRS가 높은 경우는 교차비가 6.46으로 나타났다(95% CI: 4.05-10.31). 또한 BMI가 23이상 25미만인 경우에도 GRS가 높으면 교차비가 3.13으로 나타났다(95% CI: 2.50-3.90). BMI가 25이상이고 GRS가 높은 경우를 기준으로 하였을 때 BMI가 낮아지면 교차비가 낮아지므로 BMI와 유전적 위험도 사이에 상호작용이 있음을 나타낸다.
흡연과 GRS도 상호작용이 있는 것으로 나타났으며, 비흡연이면서 GRS가 낮은 경우에 비해 GRS가 높으면서 과거흡연인 경우 교차비가 2.15(95% CI: 1.60-2.89), 현재흡연인 경우 교차비가 1.94(95% CI: 1.48-2.52)로 나타났다. 또한, 비흡연이면서 GRS가 낮은 경우에 비해 GRS가 높고 흡연량이 20갑년 이상인 경우 교차비는 2.49배 높게 나타났다(95% CI: 1.88-3.29). 음주에서도 GRS는 현재음주는 상호작용이 있는 것으로 나타났으며, 음주량에 따라서는 비음주이면서 GRS가 낮은 경우를 기준으로 음주량이 하루 10g 이상이면서 GRS가 높으면 교차비가 1.97배 높게 나타났다(95% CI: 1.51-2.57).
또한, 매운 음식을 선호하지 않으면서 GRS가 낮은 경우를 기준으로 매운 음식을 선호하고 GRS가 높으면 교차비가 1.82(95% CI: 1.51-2.19)로 높게 나타났으며, 기름진 음식을 선호하지 않으면서 GRS가 낮은 경우를 기준으로 기름진 음식을 선호하고 GRS가 높으면 교차비가 2.06(95% CI:1.62-2.61)로 나타나 음식의 선호도와 유전적 위험도 사이에 상호작용이 있는 것으로 나타났다.
논의
본 연구는 제2형 당뇨병이 발생한 대상자들과 대조군의 유전적, 생활환경적 특징을 살펴보고 이들 사이의 상호작용이 제2형 당뇨병 발생에 미치는 영향을 파악하였다.
1. 제2형 당뇨병 발생에 영향을 미치는 유전적 요인
본 연구에서는 11개의 SNPs이 제2형 당뇨병 발생과 상관성이 있는 것으로 나타나 18개의 SNPs을 제시한 선행연구[5]와 차이가 있었다. 이는 본 연구와 선행연구[5] 모두 KoGES 지역사회 코호트 데이터를 사용하였으나, 본 연구에서는 최종 추적 시까지 제2형 당뇨병이 발생한 대상자를 모두 당뇨병 환자로 선정한 반면, Go 등[5]의 연구에서는 기반자료에서 제2형 당뇨병이 있는 대상자만 포함한 것에 기인한 것으로 보인다. 즉 Go 등[5]의 연구에서 대조군은 기반조사 시점에서는 제2형 당뇨병이 발병하지 않았으나 추적과정에서 제2형 당뇨병으로 진행된 대상자들도 포함이 되었다. 따라서 당뇨병 예측 유전자들의 신뢰성 확보를 위해 한국인 집단을 대상으로 반복 연구를 진행할 필요가 있으며, 사망자 자료와 중도 탈락한 대상자들에 대한 자료를 연계 확인하여 더욱 선별된 대상자를 바탕으로 추후 연구를 진행할 필요가 있다.
연구에서 제2형 당뇨병 발생에 영향을 미치는 유전자는 PSMD6, IGF2BP2, MAEA, CDKAL1, SLC30A8, HHEX/IDE, KCNQ1, KCNJ11, MTNR1B, PEPD, HNF4A 등의 11개로, 특히 베타세포의 발달과 관련된 CDKL1, CDKN2A/B, HHEX, KCNJ11과 베타세포의 기능과 관련된 KCNQ1, SCL30A8은 다른 유전자들에 비해 교차비가 높아 임상적 활용이 높을 것으로 생각된다[22]. 각 유전자들에 대해 살펴보면, CDKL1 유전자는 6번 염색체에 위치하고 있으며 포도당 조절 및 인슐린 분비와 작용 과정에 관여하는 CDKL1 단백질을 암호화하는 기능을 한다[23]. HHEX 유전자는 10번 염색체에 위치하고 신호전달과 관련된 전사인자를 암호화하는 기능을 하며 주로 소마토스타틴 델타세포에서 발현에 관여하는 것으로 알려져 있으며, 델타 세포의 변이는 인슐린 분비 결핍과 관련된다[22, 24].
KNJ11 유전자는 11번 염색체에 위치하고 베타세포의 인슐린 분비 메커니즘 가운데 칼륨 채널을 암호화하는 기능을 하고 있으며, 이 유전자의 변이가 있는 경우 인슐린 분비가 저하되는 것으로 알려져 있다[25]. KCNQ1 유전자는 11번 염색체에 위치하며 세포막에 존재하는 전압 의존성 칼륨 채널 단백질을 암호화하여 인슐린 분비를 통해 당대사가 이루어지는 작용에 영향을 주며, Pima 인디언, 한국인, 중국인, 동아시아인, 사우디인, 유럽인에서 흔히 이 유전자의 변이가 나타난다[26]. 특히 KCNQ1 유전자는 중국인을 대상으로 한 연구에서 경구용 당뇨병 약물인 sulfonylurea의 반응에 영향을 주는 것으로 보고되었다[27]. SCL30A8은 아연수송체 단백질 ZnT8을 코딩하는 기능을 하며, 주로 인슐린 저장과 분비와 관련이 있다. SCL30A8 유전자의 발현은 췌장의 베타세포에서 가장 높게 나타나며, 제1형 당뇨병의 발병 및 합병증과도 관련이 있는 것으로 알려져 있다[28].
CDKN2A/B 유전자의 기능은 세포주기를 조절하는 p16 종양억제 인자의 억제제를 암호화하며 베타세포 기능장애와 당뇨병성 신장병증과도 관련된 것으로 보고되었으나[29], 본 연구에서는 HWE에 위배되는 것으로 나타나 한국인의 다른 집단에서 반복 연구를 통해 확인할 필요가 있다.
본 연구에서는 제2형 당뇨병과 관련 있는 11개의 SNPs을 이용하여 조우성 모델에 따라 유전적 요인을 GRS로 계산하였다. 실제로 GRS는 제2형 당뇨군과 대조군에서 차이가 나타났으므로 유전적 요인을 점수로 표현하는데 신뢰성 있는 결과라 할 수 있다. 제2형 당뇨병과 같이 여러 유전자들이 질병 발생에 관여하면서 각각의 유전자들이 낮은 교차비를 가지는 경우, 관련된 유전자 전체를 이용하여 GRS를 계산하여 유전과 환경의 상호작용을 설명하는 것은 유용한 전략으로 사용되고 있다[30]. 그러나 한국인을 대상으로 제2형 당뇨병 발생의 유전적 요인을 예측할 수 있는 GRS를 확인하는 연구가 거의 보고된 바가 없어 본 연구의 결과와 비교할 수 있는 추후 연구가 필요하다. 또한 이러한 제2형 당뇨병의 유전적인 요인들의 복합적인 효과를 나타내는 GRS가 유전적 요인을 대표하는데 신뢰도를 높이기 위해서 한국인에게 특별히 감수성이 나타나는 유전자를 찾기 위한 노력이 계속되어야 할 것이다.
2. 제2형 당뇨병 발생에 영향을 미치는 요인
제2형 당뇨병 발생에 영향을 미치는 요인을 확인하기 위하여 Cox 비례위험모형을 통해 살펴본 결과 다변량 분석의 최종 모형에서 BMI, GRS, 흡연, 매운 음식 선호도가 유의한 것으로 나타났다. 유전적인 요인은 BMI처럼 변경 가능한 요인은 아니지만 제2형 당뇨병 발생의 예측요인으로 사용될 수 있어 유전적 고위험군 대상자들을 선별하는 것이 제2형 당뇨병 예방에서 중요하다[31]. 그러나 제2형 당뇨병은 많은 유전자의 변이가 복합적으로 관여하기 때문에 이를 통합해서 유전적 위험도를 계산하기가 어렵고[32] 인종과 지역에 따라 다른 유전적 변이를 가지고 있으므로 공통적인 GRS로 일반화해서 적용할 수 없다[22,23,33]. 따라서 그 인구집단의 유전적인 요인을 잘 나타낼 수 있는 유전적 변이를 찾아낼 필요가 있으나 한국인에서 제2형 당뇨병을 예측할 수 있는 유전적 변이에 대한 연구가 아직까지 많이 이루어지지 않았으므로 먼저 제2형 당뇨를 잘 예측할 수 있는 유전적 변이를 찾아내고 이를 바탕으로 유전적 위험을 검증할 수 있는 도구가 마련되어야 할 것이다.
3. 제2형 당뇨병 발생에서 유전과 생활환경의 상호작용
본 연구에서는 GRS와 상호작용을 하는 생활환경 요인은 BMI, 매운 음식 선호도, 기름진 음식의 선호도, 음주, 흡연으로 나타났다. BMI는 유전과의 상호작용이 매우 높게 나타났는데 특히 비만의 기준에 해당되는 25kg/m2이상에서는 유전적 요인이 높은 경우에는 제2형 당뇨병의 발생 위험이 6.46배까지 상승하였다. 비만은 제2형 당뇨병 발생의 가장 큰 위험요인으로 보고되고 있으며 제2형 당뇨병환자의 60-90%가 비만이고, 비만을 발생시키는 고열량식이와 신체활동의 저하는 제2형 당뇨병 발생에 밀접한 관련이 있다고 알려져 있다[34]. 따라서 유전적 위험도가 높은 사람에게 정상 BMI를 유지하는 것이 제2형 당뇨병 예방을 위해 중요하다.
본 연구에서 흡연은 BMI 다음으로 유전과 상호작용이 높은 것으로 나타났고 유전적 위험이 높을 경우 20갑년 미만인 경우 1.78배, 20갑년 이상인 경우 2.49배로 높게 나타났다. 흡연은 혈관질환과 사망률을 증가시키는 위험인자로 알려져 있을 뿐 아니라[35] 당뇨병의 발생에도 영향을 미치는 요인으로 알려져 있어[36] 본 연구에서 흡연이 위험요인으로 나타난 것과 일치하는 결과이다. 또한 유전적 위험도와 음주가 상호작용이 있는 것으로 나타났는데 알코올 섭취는 제2형 당뇨병 발생의 위험인자로 알려져 있으며[37] 본 연구의 결과 음주량에 상관없이 음주여부 자체로도 유전과 상호작용을 통해 위험이 높은 것으로 나타났다. 그러나 당뇨병 예방중재에 대한 선행 연구를 살펴보면 대부분 에너지 섭취와 신체활동에 대해서 강조하고 있어[38], 본 연구결과에서 나타난 흡연과 음주에 대해서는 적극적으로 중재에 포함하고 있지 않다. 따라서 제2형 당뇨병 발생의 유전적 고위험군을 선별하여 맞춤형 예방중재 시 금연과 금주를 프로그램에 포함시킬 필요가 있다.
식사 선호도에서 매운 음식과 기름진 음식을 선호도는 유전적 위험이 높은 경우에 제2형 당뇨병 발생위험이 높아졌다. 실제 비만한 사람일수록 매운맛에 대한 선호도가 높고, 당뇨병으로 진단받은 사람들의 과거 식습관을 역추적했을 때 기름진 음식의 선호도가 높았으므로[39], 맵고 기름진 음식은 비만과 제2형 당뇨병의 위험요인이라고 볼 수 있다. 본 연구에서 GRS가 낮은 경우에는 맵고 기름진 음식의 선호도가 있어도 제2형 당뇨병에 대한 위험이 높아지지 않았으나 GRS가 높은 경우에는 맛 선호도와의 상호작용으로 인해 제2형 당뇨병 발생에 위험이 높게 나타났다. 특히 매운 음식은 인스턴트 식단과 한국인의 식단에서 주로 나타나는 특징이므로 본 연구의 결과는 제2형 당뇨병예방을 위하여 한국인의 식단에 있어서 식이지도의 필요성을 보여주고 있다.
본 연구는 제2형 당뇨병 발생의 유전과 환경 역할 모델[2]을 바탕으로 제2형 당뇨병 발생에 있어서 원인이 되는 유전적인 요인과 다양한 생활환경 요인들을 구체적으로 확인하였다는데 의의가 있다. 지금까지 제2형 당뇨병에서 유전과 환경의 상호작용을 조사한 선행연구들은 생활환경 요인으로 섭취영양성분과 같이 일부 요인만 분석하거나[11], 당화혈색소나 혈당수치와 같은 임상지표들을 포함하여 제2형 당뇨병에 영향을 미치는 생활환경 요인을 포괄적으로 밝혀내지 못하였다[40]. 또한, 유전적 요인과 일부 생활환경 요인은 민족에 따라 차이가 있으며, Pima 인디언과 같이 유전과 환경의 상호작용이 그 민족만의 특별한 질병 감수성을 나타낼 수 있으므로[26] 본 연구에서는 한국인의 제2형 당뇨병 발생에 영향을 미치는 유전적, 생활환경 요소를 파악하고 상호작용의 특징을 파악하였다는 점에도 의의가 있다. 특히 식생활에 있어서 짠 음식, 매운 음식, 기름진 음식에 대한 한국형 정의를 확립하여 유전적으로 위험도가 높은 사람에게 제2형 당뇨병을 예방할 수 있는 식이 프로그램을 제공할 필요가 있다.
본 연구의 주요 제한점은 기반자료에서 6차 추적까지 제2형 당뇨병의 누적 발생을 포함하였으나 기반자료를 바탕으로 분석하였기 때문에 조사시점에서 당뇨병 발생시점까지 환경적인 요인의 변화 과정을 분석에 포함하지 못하는 점이다. 특히 우울이나 스트레스와 같은 심리 사회적인 요인들은 시간의 흐름에 따라 변화가능성이 높기 때문에 더 정확한 영향을 살펴보기 위해서는 추적자료의 분석이 필요한 것으로 생각된다.
결론
본 연구에서 제2형 당뇨병 발생과 관련된 요인으로 11개의 SNPs과 GRS, BMI, 흡연, 매운 음식 선호도였다. GRS와 상호작용이 있는 생활환경 요인으로는 BMI, 매운 음식 및 기름진 음식의 선호도, 음주, 흡연으로 나타났다. 따라서 유전적인 위험이 높은 경우에는 환경적 위험 요인에 노출될 때 제2형 당뇨병의 위험이 훨씬 더 높아질 수 있으므로 유전적 고위험 대상자들을 선별하여 개별화된 중재를 시행할 필요가 있으며 특히 체중감소, 식사습관, 음주, 흡연의 위험을 강조하는 중재가 제공되어야 할 것이다.
본 연구 결과 다음과 같이 제언하고자 한다. 첫째, 본 연구에서 유의하게 나타난 11개의 SNPs과 제2형 당뇨병과의 관련성을 재확인하기 위하여 추가적으로 한국인 집단에서 반복 연구가 이루어져야 하며, 둘째, 추적 데이터의 자료를 이용하여 환경적 요인들의 변화와 차이를 확인할 수 있는 후속 연구를 제언한다. 셋째, 유전적 위험도에 따라 BMI, 식사습관, 음주, 흡연을 포함한 제2형 당뇨병 예방중재를 개발하여 효과를 확인할 수 있는 연구를 제언한다.
Notes
CONFLICT OF INTEREST
The authors declared no conflict of interest.
AUTHORSHIP
SH and SJ contributed to the conception and design of this study; SH collected data; SH and SJ performed the statistical analysis and interpretation; SH and SJ drafted the manuscript; SH and SJ critically revised the manuscript; SH and SJ supervised the whole study process. All authors read and approved the final manuscript.
FUNDING
None.
Supplementary Materials
Odds Ratios and Genetic Risk Scores of 18 SNPs Associated with T2D