л…јмқҳ
ліё м—°кө¬лҠ” 비л§Ңмқҙ л°ңмғқн•ҳмҳҖмқ„ л•Ң мқјм–ҙлӮҳлҠ” лҢҖмӮ¬м„ұ ліҖнҷ”лҘј ліҙкё° мң„н•ҳм—¬ мӮ¬лһҢкіј к°ҖмһҘ мң мӮ¬н•ҳкІҢ лӮҳнғҖлӮҳлҠ” C57BL/6J л§Ҳмҡ°мҠӨм—җкІҢ ліҙнҺём ҒмңјлЎң мӮ¬мҡ©н•ҳлҠ” 45%мҷҖ 60% кі м§Җл°©мӢқмқҙлҘј к°Ғк°Ғ кіөкёүн•ҳм—¬ 10%мқҳ м§Җл°©мқ„ н•Ёмң н•ҳкі мһҲлҠ” м •мғҒмӢқмқҙ(CD)лҘј кіөкёүн•ҳмҳҖмқ„ л•ҢмҷҖ м–ҙл–Ө ліҖнҷ”к°Җ мқјм–ҙлӮҳлҠ”м§Җ 비көҗ, 분м„қн•ҳмҳҖлӢӨ[
13]. м§Җл°©н•Ёмң лҹүмқҙ лҶ’кұ°лӮҳ кіјлҸ„н•ң м№јлЎңлҰ¬ м„ӯм·Ём—җ мқҳн•ҙ 비л§Ңмқҙ л°ңмғқн•ҳл©ҙ м§Җл°©лҢҖмӮ¬мқҳ мһҘм• к°Җ мқјм–ҙлӮҳл©ҙм„ң к°„мЎ°м§Ғм—җ кіјлҸ„н•ң м§Җл°©мқҙ 축м Ғмқҙ лҗҳлҠ” м§Җл°©к°„мҰқ лҳҗлҠ” 비м•ҢмҪңм„ұ м§Җл°©к°„м§Ҳнҷҳмқҳ л°ңмғқмқҙ мҰқк°Җн•ҳкІҢ лҗңлӢӨ. м§Җл°©к°„мқҳ мҰқк°Җ кё°м „мқҖ мІ«м§ё, мӢқмқҙ м§Җл°© нҳ№мқҖ м№јлЎңлҰ¬мқҳ кіјлӢӨ м„ӯм·Ём—җ мқҳн•ң к°„мңјлЎң м§Җл°© мң мһ…(inflow)мқҳ мҰқк°Җ, л‘ҳм§ё, м§Җл°©мЎ°м§Ғ(adipose tissue)м—җм„ң м§Җ방분н•ҙ(lipolysis)мқҳ мҰқк°ҖлЎң мқён•ң к°„мңјлЎңмқҳ кіјлҸ„н•ң мң лҰ¬м§Җл°©мӮ°(free fatty acids)мқҳ мң мһ…, м…Ӣм§ё, к°„м—җм„ң мӢ к·ң м§Җл°©н•©м„ұ(de novo lipogenesis)мқҳ мҰқк°Җ, л„·м§ё, к°„м—җм„ң мҙҲм Җл°ҖлҸ„мҪңл ҲмҠӨн…ҢлЎӨ(Very Low Density Lipoprotein)мқ„ нҶөн•ң TG мң м¶ң(outflow)мқҳ к°җмҶҢ, к·ёлҰ¬кі лӢӨм„Ҝм§ёлЎң к°„м—җм„ң м§Җл°©мӮ° мӮ°нҷ”(oxidation)мқҳ к°җмҶҢ, мҰү кіөліө мӨ‘ лӢ№мӢ мғқ(gluconeogenesis) л“ұмқҳ мЈјмҡ” м—җл„Ҳм§Җмӣҗ мӮ¬мҡ©мқҳ к°җмҶҢ л“ұмқҙлӢӨ[
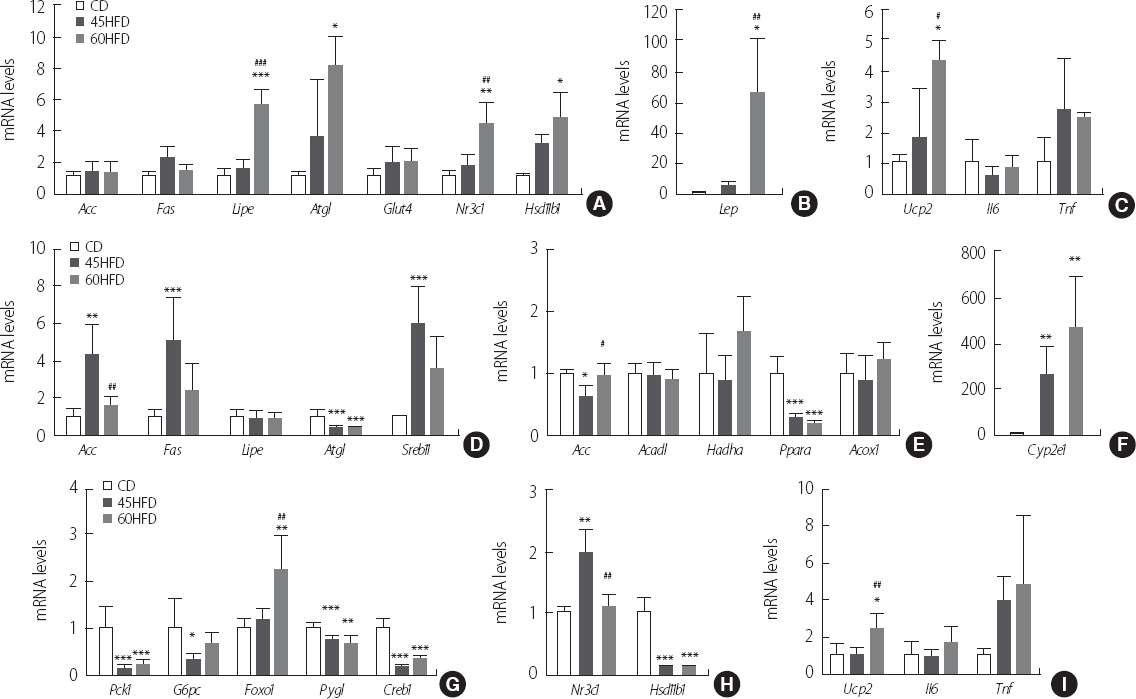
14]. ліё м—°кө¬лҘј нҶөн•ҙ лӮҳнғҖлӮң кІ°кіјл“ӨмқҖ м§Җл°©к°„мқҙ л°ңмғқн•ҳлҠ” кё°м „мқ„ мҲңм°Ём ҒмңјлЎң мҰқлӘ…н•ҳкі мһҲлӢӨ. 45% лҳҗлҠ” 60%мқҳ кі м§Җл°©мӢқмқҙлҘј кіөкёүн•ҳкі 8мЈјк°Җ м§ҖлӮң мӢңм җм—җ м§Җл°©мЎ°м§Ғм—җм„ңлҠ” м§Җл°©мқҳ н•©м„ұліҙлӢӨлҠ” м§Җл°© 분н•ҙм—җ кҙҖм—¬н•ҳлҠ” нҡЁмҶҢл“Ө,
LipeмҷҖ
Atglмқҳ л°ңнҳ„мқҙ мң мқҳн•ҳкІҢ мҰқк°Җн•ҳмҳҖлӢӨ.
LipeмҷҖ
AtglмқҖ м§Җл°©мЎ°м§Ғм—җм„ң нҠёлҰ¬кёҖлҰ¬м„ёлҰ¬л“ңлҘј лӢӨмқҙм•„мӢӨкёҖлҰ¬м„ёлЎӨ(diacylglycerol;DAG)кіј мң лҰ¬м§Җл°©мӮ°(free fatty acids)мңјлЎң 분н•ҙн•ҳлҠ” нҡЁмҶҢмқҙлӢӨ. лҸҷмӢңм—җ к°„м—җм„ңлҠ” м§Җл°©мғқм„ұмқҳ м „мӮ¬мҡ”мҶҢ(transcription factor)мқё
Srebf1мқ„ нҶөн•ң
Fas,
AccмҷҖ к°ҷмқҖ м§Җм§Ҳ н•©м„ұ нҡЁмҶҢмқҳ л°ңнҳ„мқҙ мҰқк°Җн•ҳкі к·ё кІ°кіјлЎң нҳҲмӨ‘ м§Җм§Ҳ м§Җн‘ңмқҳ мҰқк°Җк°Җ мқјм–ҙлӮЁмқ„ нҷ•мқён•ҳмҳҖлӢӨ. м •мғҒм Ғмқё лҢҖмӮ¬кіјм •м—җм„ң мқёмҠҗлҰ°мқҖ м§Җл°©мЎ°м§Ғм—җм„ң м§Җл°© 분н•ҙлҘј м–өм ңн•ҳм§Җл§Ң ліё м—°кө¬м—җм„ңмҷҖ 60HFDкө°м—җм„ңлҠ” нҳҲмӨ‘ мқёмҠҗлҰ°мқҖ мҰқк°Җн•ҳлӮҳ мқёмҠҗлҰ° м Җн•ӯм„ұмқҙ мҲҳл°ҳлҗҳл©ҙм„ң м§Җл°©мЎ°м§Ғмқҳ м§Җм§Ҳ 분н•ҙк°Җ мҙү진лҗҳкі (
Fig. 2A), 분н•ҙлҗң мң лҰ¬м§Җл°©мӮ°мқҖ к°„мңјлЎң мң мһ…лҗҳлҠ” лҸҷмӢңм—җ
Srebf1мқҳ мң м „мһҗ л°ңнҳ„мқ„ мҰқк°ҖмӢңнӮӨлҠ” кІғмқ„ нҷ•мқён• мҲҳ мһҲм—ҲлӢӨ[
15]. мқҙлҹ¬н•ң мқёмҠҗлҰ° м Җн•ӯм„ұкіј к°„м—җм„ң мӨ‘м„ұм§Җл°©мқҳ 축м Ғм—җ лҢҖн•ң
Srebf1мқҳ м—ӯн• мқҖ лӢӨлҘё лҸҷл¬јлӘЁлҚёмқ„ нҶөн•ҙм„ңлҸ„ мҰқлӘ…лҗҳм—ҲлӢӨ. кі лҸ„비л§Ңкіј мқёмҠҗлҰ° м Җн•ӯм„ұмқҳ нҠ№м§•мқё
ob/ob miceм—җм„ң м§Җл°©к°„ лі‘ліҖмқҙ кҙҖм°°лҗҳм—Ҳкі ,
ob/ob miceмқҳ
Srebf1 мң м „мһҗлҘј 비нҷңм„ұнҷ” мӢңмј°мқ„ л•Ң к°„мЎ°м§Ғм—җ мӨ‘м„ұм§Җл°©мқҳ 축м Ғмқҙ м•Ҫ 50% м •лҸ„ к°җмҶҢн•Ёмқ„ ліҙкі н•ң м—°кө¬лҘј нҶөн•ҙ
Srebf1к°Җ мқёмҠҗлҰ° м Җн•ӯм„ұлҸҷл¬ј лӘЁлҚём—җм„ң м§Җл°©к°„мқҳ л°ңмғқм—җ мӨ‘мҡ”н•ң м—ӯн• мқ„ лӢҙлӢ№н•Ёмқ„ м•Ң мҲҳ мһҲлӢӨ[
16].
мң лҰ¬м§Җл°©мӮ°мқҖ лІ нғҖ мӮ°нҷ”лҘј нҶөн•ҳм—¬ м—җл„Ҳм§ҖмӣҗмңјлЎң м „нҷҳлҗҳм–ҙм•ј н•ҳм§Җл§Ң ліё м—°кө¬мқҳ кІ°кіјмҷҖ к°ҷмқҙ м§ҖлӮҳм№ң м§Җл°©мқҳ 축м ҒмқҖ лІ нғҖмӮ°нҷ”лҘј м–өм ңн•ҳкі (
Fig. 2E)
Cyp2e1мқҳ л°ңнҳ„мқ„ мҰқк°ҖмӢңмјң лҢҖмІҙ мӮ°нҷ”кіјм •мқ„ мқјмңјнӮӨкІҢ н•ңлӢӨ(
Fig. 2F). к°„м„ёнҸ¬мқҳ м§Җл°©мӮ° мӮ°нҷ”нҡЁмҶҢмқё
Cyp2e1мқҖ нҷңм„ұмқҙ мҰқк°Җн•ҳл©ҙ к°„м—җ 축м Ғлҗң мң лҰ¬м§Җл°©мӮ°мңјлЎңл¶Җн„° нҷңм„ұмӮ°мҶҢмў…мқ„ мғқм„ұмӢңмјң мӮ°нҷ”мҠӨнҠёл ҲмҠӨлҘј мқјмңјнӮӨлҠ” кІғмңјлЎң м•Ңл Өм ёмһҲлӢӨ[
17]. м„ёнҸ¬мқҳ лҢҖмӮ¬кіјм •м—җм„ң лҜёнҶ мҪҳл“ңлҰ¬м•„лҠ” ATPлҘј мғқм„ұн•ҳлҠ” кё°лҠҘмқ„ лӢҙлӢ№н•ңлӢӨ. мқҙ кіјм • мӨ‘м—җ лҜёнҶ мҪҳл“ңлҰ¬м•„лҠ” мғқмЎҙм Ғ л°ҳмқ‘мңјлЎң мӮ°мҶҢлЎң м „мһҗмқҳ мқҙлҸҷмқҙ мҰқк°Җлҗҳм–ҙ нҷңм„ұмӮ°мҶҢмў…(reactive oxygen species, ROS)мқҙ мғқм„ұн•ҳлҠ”лҚ°, м§ҖлӮҳм№ң ROSлҠ”
IL-6мҷҖ
TNFлҘј нҸ¬н•Ён•ң м—¬лҹ¬ мӮ¬мқҙнҶ м№ҙмқёмқҳ м „мӮ¬мқёмһҗмқё NF-kBлҘј нҷңм„ұнҷ”мӢңмјң лҜёнҶ мҪҳл“ңлҰ¬м•„мқҳ кё°лҠҘм Җн•ҙмҷҖ ROS мғқм„ұмқ„ мҰқк°ҖмӢңнӮӨкІҢ лҗңлӢӨ. лҜёнҶ мҪҳл“ңлҰ¬м•„ л§үм—җ мЎҙмһ¬н•ҳлҠ”
UCP2лҠ” мқҙлҹ¬н•ң ROSмқҳ мғқм„ұмқ„ м–өм ңн•ҳлҠ” лӢЁл°ұм§ҲлЎң 비м•ҢмҪңм„ұ м§Җл°©к°„м§Ҳнҷҳ (non-alcoholic fatty liver disease, NAFLD)кіј 비л§Ңм—җм„ң мӮ°нҷ”мҠӨнҠёл ҲмҠӨ(oxidative stress)к°Җ лҶ’мқ„ л•Ң л°ңнҳ„мқҙ мҰқк°Җн•ңлӢӨкі ліҙкі лҗҳм–ҙмһҲлӢӨ[
18]. ліё м—°кө¬м—җм„ңлҸ„ 16мЈјлҸҷм•Ҳ 45%мқҳ кі м§Җл°©мӢқмқҙлҘј м Ғмҡ©н•ң мғқмҘҗмқҳ м§Җл°©мЎ°м§Ғм—җм„ң
Ucp2мҷҖ мӮ¬мқҙнҶ м№ҙмқё(
IL6,
Tnf)мқҳ мҰқк°ҖлҘј нҷ•мқён• мҲҳ мһҲм—ҲлҠ”лҚ°(
Fig. 3C), мқҙлҠ” м§Җл°©мӮ°м—җм„ң мғқм„ұлҗң нҷңм„ұмӮ°мҶҢмқҳ мҰқк°Җм—җ л”°лқј мҰқк°Җн•ң мқјл Ёмқҳ л°ҳмқ‘мқҙлқј ліј мҲҳ мһҲлӢӨ.
ліё м—°кө¬м—җм„ң
Lep,
Nr3c1 к·ёлҰ¬кі
Hsd11b1 м„ё к°Җм§Җ мң м „мһҗлҠ” мӢқмқҙмқҳ кі м§Җл°© н•Ёлҹүм—җ л”°лқј л°ңнҳ„мқҳ м°ЁмқҙлҘј ліҙмҳҖлӢӨ.
LepмқҖ м§Җл°©м„ёнҸ¬м—җм„ң 분비лҗҳлҠ” нҳёлҘҙлӘ¬ мң м „мһҗлЎң м •мғҒмӢқмқҙлҘј н•ҳлҠ” л§Ҳмҡ°мҠӨм—җ 비н•ҙ 45HFDкө°м—җм„ң м•Ҫ 6л°°, 60HFDкө°м—җм„ңлҠ” м•Ҫ 21.5л°°мқҳ mRNA л°ңнҳ„мқҳ мҰқк°ҖлҘј ліҙмқёлҚ° 비н•ҙ мӢқмқҙ м„ӯм·Ёлҹүм—җлҠ” нҒ° ліҖнҷ”лҘј ліҙмқҙм§Җ м•ҠмқҖ кІғмңјлЎң ліҙм•„ м§Җл°©лҹүмқҳ мҰқк°ҖлҘј к°„м ‘м ҒмңјлЎң лӮҳнғҖлӮҙкё°лҸ„ н•ҳл©°[
19,
20], 비л§Ңкө°м—җм„ң нқ”нһҲ лӮҳнғҖлӮҳлҠ” л ҷнӢҙ м Җн•ӯм„ұмқҳ кІ°кіјлЎңлҸ„ н•ҙм„қн• мҲҳ мһҲлӢӨ. 60HFDкө°м—җм„ң мӮ¬лЈҢм„ӯм·Ёлҹүмқҙ м•Ҫ 20% к°җмҶҢн•ҳмҳҖмңјлӮҳ мӢӨм ң л ҷнӢҙмқҳ м Җн•ӯм„ұмқҙ мһҲлҠ”м§ҖлҠ” л ҷнӢҙ мҲҳмҡ©мІҙлҘј нҸ¬н•Ён•ҳлҠ” кҙҖл Ё нҳёлҘҙлӘ¬мқҳ л°ңнҳ„кіј 분비м—җ лҢҖн•ң нӣ„мҶҚ мЎ°мӮ¬к°Җ н•„мҡ”н• кІғмңјлЎң ліҙмқёлӢӨ. лҳҗн•ң, кёҖлЈЁмҪ”мҪ”лҘҙнӢ°мҪ”мқҙл“ңлҠ” 비л§Ңкіј 비м•ҢмҪңм„ұ м§Җл°©к°„м§Ҳнҷҳм—җ м—°кҙҖм„ұмқҙ мһҲлҠ” кІғмңјлЎң м•Ңл ӨмЎҢлӢӨ. Andrews л“ұмқҳ м—°кө¬м—җ мқҳн•ҳл©ҙ л¶Ҳнҷңм„ұнҳ• мҪ”лҘҙнӢ°мҶҗ(cortisone)мқ„ нҷңм„ұнҳ• мҪ”нӢ°мҶ”(cortisol)лЎң ліҖнҷҳмӢңнӮӨлҠ” нҡЁмҶҢмқё
Hsd11b1лҘј м–өм ңн•ҳл©ҙ мІҙмӨ‘ л°Ҹ нҳҲмӨ‘ м§Җм§ҲмҲҳм№ҳмқҳ к°җмҶҢнҷ” н•Ёк»ҳ к°„мқҳ мқёмҠҗлҰ° м Җн•ӯм„ұмқҙ лӮ®м•„진лӢӨкі ліҙкі н•ҳмҳҖлӢӨ[
21]. мқҙлҠ” ліё м—°кө¬кІ°кіјм—җм„ңлҸ„ мң мӮ¬н•ҳкІҢ лӮҳнғҖлӮҳ кі м§Җл°©мӢқмқҙлҘј н• мҲҳлЎқ м§Җл°©м„ёнҸ¬мқҳ
Hsd11b1мқҳ л°ңнҳ„кіј н•Ёк»ҳ мҲҳмҡ©мІҙмқё
Nr3c1 мқҳ л°ңнҳ„лҸ„ мҰқк°Җн•ҳлҠ” кІғмқ„ ліј мҲҳ мһҲм—ҲлӢӨ. к·ёлҹ¬лӮҳ к°„мЎ°м§Ғм—җм„ң
Hsd11b1мқҳ л°ңнҳ„мқҖ нҳ„м Җн•ҳкІҢ к°җмҶҢн•ҳкі (
Fig. 3H), нҳҲмӨ‘ мҪ”лҘҙнӢ°мҪ”мҠӨн…ҢлЎ мқҳ мҲҳм№ҳм—җлҠ” ліҖнҷ”к°Җ м—ҶлҠ” кІғмңјлЎң ліј л•Ң, м „мӢ м Ғмқё нҳҲм•Ў лӮҙ кёҖлЈЁмҪ”мҪ”лҘҙнӢ°мҪ”мқҙл“ңмҷҖ мЎ°м§Ғм—җм„ң л°ңнҳ„лҗҳлҠ” нҳёлҘҙлӘ¬мқҳ м–‘мқҖ лҸҷмқјн•ҳм§Җ м•ҠлӢӨлҠ” м җмқ„ м•Ң мҲҳ мһҲлӢӨ. лҳҗн•ң к°Ғ мЎ°м§Ғл§ҲлӢӨ л°ңнҳ„мқҳ м •лҸ„к°Җ лӢӨлҘҙлҜҖлЎң мқҙ нҳёлҘҙлӘ¬м—җ мқҳн•ң лҢҖмӮ¬кё°лҠҘмқҳ мЎ°м Ҳм—җ лҢҖн•ҙ м•һмңјлЎң лҚ” м—°кө¬лҗҳм–ҙм•ј н• кІғмқҙлӢӨ. лҳҗн•ң, мқёмҠҗлҰ°м Җн•ӯм„ұмқҖ 비л§ҢмқҙлӮҳ м ң2нҳ•лӢ№лҮЁмқҳ м Җл“ұкёү м—јмҰқ(low-grade inflammation) мғҒнғңм—җ мқҳн•ҙ лҸҷл°ҳлҗҳм–ҙ м—јмҰқ мӮ¬мқҙнҶ м№ҙмқёмқ„ 분비мӢңнӮӨкі ,
11ОІHSD1мқ„ мҰқк°ҖмӢңмјң кёҖлЈЁмҪ”мҪ”лҘҙнӢ°мҪ”мқҙл“ңлҘј нҷңм„ұнҷ”мӢңнӮЁлӢӨ[
22].
TNF лҳҗлҠ” dexamethasoneмқҙ мҘҗмқҳ м§Җл°©м„ёнҸ¬м—җм„ң мқёмҠҗлҰ°м Җн•ӯм„ұмқ„ мң лҸ„н•ңлӢӨкі ліҙкі лҗҳм—ҲлӢӨ[
23].
Foxo1мқҖ мқёмҠҗлҰ° мӢ нҳём „лӢ¬мқ„ л§Өк°ңн•ҳлҠ” м „мӮ¬мЎ°м ҲмқёмһҗлЎң мқёмҠҗлҰ° мҲҳмҡ©мІҙлҘј м ңкұ°н•ң мң м „мһҗліҖнҳ• мҘҗмқҳ к°„м—җм„ң мҰқк°Җлҗҳм—Ҳкі ,
Foxo1мқ„ м ңкұ°н•ҳл©ҙ лҜёнҶ мҪҳл“ңлҰ¬м•„мқҳ мӮ°нҷ” мӢ 진лҢҖмӮ¬мҷҖ мғқн•©м„ұ кё°лҠҘмқ„ нҡҢліөмӢңмј°лӢӨ[
24]. ліё м—°кө¬ кІ°кіјм—җм„ң лӮҳнғҖлӮң
Foxo1мқҳ мҰқк°ҖлҠ” лӢ№лҢҖмӮ¬мҷҖлҠ” мғҒкҙҖм—Ҷмқҙ лҜёнҶ мҪҳл“ңлҰ¬м•„мқҳ кё°лҠҘмқ„ лӮ®м¶”лҠ” кІғмңјлЎң ліҙмқёлӢӨ.
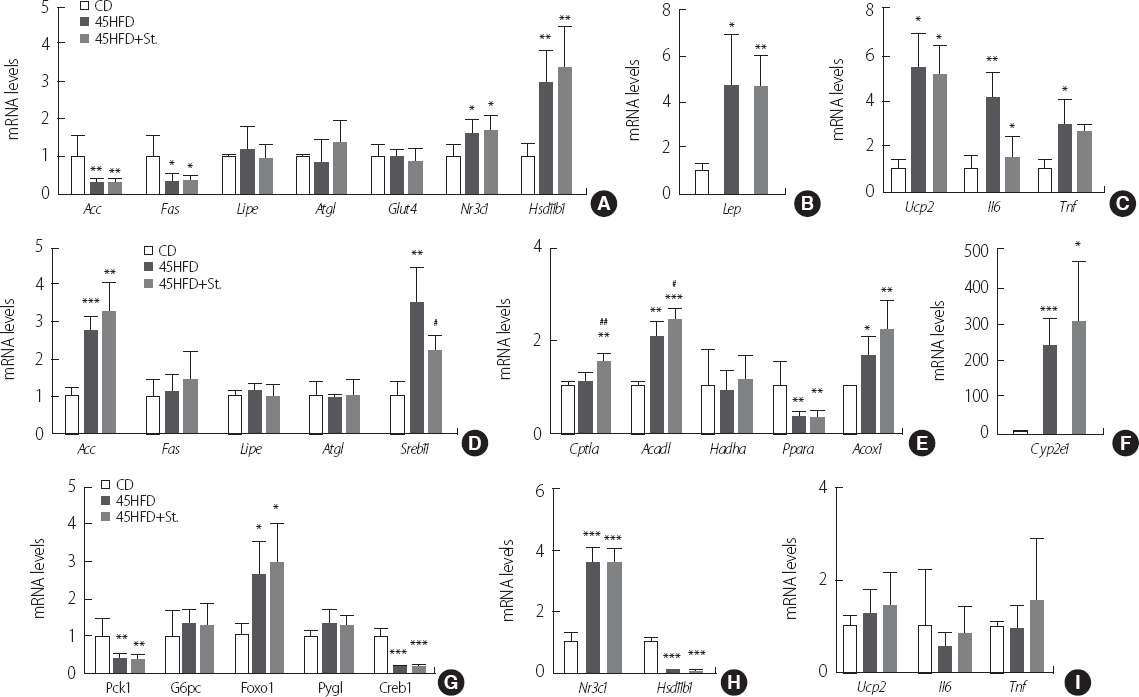
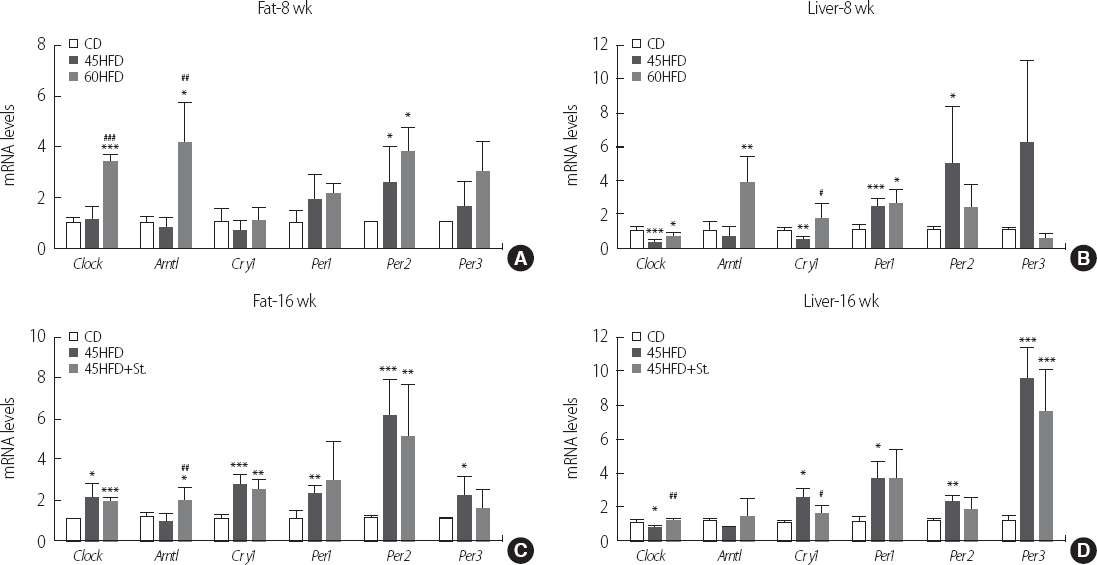
ліё м—°кө¬м—җм„ң кі м§Җл°©мӢқмқҙлҘј кіөкёүн•ң л§Ҳмҡ°мҠӨл“Өмқҳ м§Җл°©кіј к°„ мЎ°м§Ғмқҳ мғқмІҙмӢңкі„ мң м „мһҗл“Өмқҳ л°ңнҳ„мқҖ м§Җл°© н•Ёлҹүм—җ л”°лқј мҰқк°Җн•ҳлҠ” нҢЁн„ҙмқ„ ліҙмҳҖлӢӨ. лҳҗн•ң 16мЈјл № 45HFDкө°мқҳ нҳҲмӨ‘ мғқнҷ”н•ҷ м§Җн‘ңк°Җ 8мЈјл № 60HFDкө°мқҳ м§Җн‘ңл“Өл§ҢнҒј мғҒмҠ№н•ҳмҳҖлҠ”лҚ° мғқмІҙмӢңкі„ мң м „мһҗл“Өмқҳ л°ңнҳ„ м •лҸ„к°Җ лҸҷмқјн•ҳкІҢ мғҒмҠ№лҗҳлҠ” кІғмқ„ нҷ•мқён• мҲҳ мһҲм—ҲлӢӨ. лӢЁ, 60HFDкө°мқҳ к°„мЎ°м§Ғкіј 16мЈјл № 45HFDкө° м§Җл°©мЎ°м§Ғмқҳ
Per2,
3мқҳ мң м „мһҗ л°ңнҳ„мқҙ нҳ„м Җн•ҳкІҢ к°җмҶҢн•ҳлҠ” кІғмңјлЎң ліҙм•„ мң м „мһҗл“Өмқҳ м•„мқҙмҶҢнҳ•(isoform)л“Өмқҙ к°Ғк°Ғ лӢӨлҘё кё°лҠҘмқ„ к°Җм§Җкі мһҲлҠ” кІғмңјлЎң ліҙмқҙл©° мЎ°м§Ғм—җ л”°лқј мң м „мһҗмқҳ л°ңнҳ„нҢЁн„ҙкіј мЎ°м Ҳмқёмһҗк°Җ лӢӨлҘј кІғмқ„ мҳҲмғҒн• мҲҳ мһҲлӢӨ. м§Җл°©м„ёнҸ¬мқҳ мЈјмҡ” м—…л¬ҙлҠ” м§Җл°©лҢҖмӮ¬мҷҖ м—јмҰқ мӮ¬мқҙнҶ м№ҙмқёмқҳ 분비мқҙл©°,
Clockкіј
ArntlмқҖ кі м§Җл°©мӢқмқҙм—җ мқҳн•ң м§Җл°©лҢҖмӮ¬мқҳ нҷңм„ұмқ„ мЎ°м Ҳн•ҳлҠ” кІғмңјлЎң мғқк°ҒлҗңлӢӨ[
25].
Clockкіј
Arntlмқҳ нҷңм„ұмңјлЎң мҰқк°Җлҗң
CryмҷҖ
Per л°ңнҳ„мқҳ мҰқк°ҖлҠ” negative feedbackм—җ мқҳн•ҳм—¬
Clockкіј
Arntlмқҳ л°ңнҳ„мқ„ м–өм ңн•ҳлҠ” нҢЁн„ҙмқ„ ліҙмҳҖлӢӨ.
кі м§Җл°©мӢқмқҙлЎң мң лҸ„н•ҳлҠ” 비л§Ңм—°кө¬лҘј н• л•Ң 60%мқҳ кі м§Җл°© мӢқмқҙлҠ” 짧мқҖ мӢңк°„ лӮҙм—җ 비л§Ңмқ„ мң лҸ„н• мҲҳ мһҲм–ҙ мқјл°ҳм ҒмңјлЎң м“°мқҙлҠ” л°©лІ•мқҙлӮҳ 1 g лӢ№ 5.24 kcalлЎң 232 mgмқҳ мҪңл ҲмҠӨн…ҢлЎӨмқ„ н•Ёмң н•ҳкі мһҲм–ҙ мӮ¬лһҢмқҳ мӢқмқҙм—җ кІ¬мЈјм–ҙ ліҙл©ҙ нҳ„мӢӨм Ғмқҙлқј ліҙкё° м–ҙл өкі [
9] 비л§Ң л°ңмғқмқҳ м •лҸ„к°Җ мқҙлҜё н•ңкі„м җмқ„ лҸ„лӢ¬н•ң мғҒнғңмқҙлҜҖлЎң ліё м—°кө¬м—җм„ңлҠ” 45% кі м§Җл°©мӢқмқҙлЎң мӨ‘к°„ м •лҸ„мқҳ 비л§Ңмқ„ мң лҸ„н•ҳкі мқҙлҘј мҠӨнҠёл ҲмҠӨк°Җ к°ҖмҶҚмӢңнӮ¬ мҲҳ мһҲлҠ”м§Җ ліҙкі мһҗ н•ҳмҳҖлӢӨ. л¶ҖлҸҷ мҠӨнҠёл ҲмҠӨ(immobilization stress) лӘЁлҚёмқҖ л§Ңм„ұ мҠӨнҠёл ҲмҠӨлҘј м Ғмҡ©н•ҳлҠ”лҚ° мһҗмЈј м“°мқҙлҠ” л°©лІ•мңјлЎң мӢңмғҒн•ҳл¶Җ-лҮҢн•ҳмҲҳмІҙ-л¶ҖмӢ 축(hypothalamus-pituitary-adrenal axis)кіј көҗк°җмӢ кІҪкі„(sympathetic nervous system)мқҳ нҷңм„ұнҷ”лҘј нҠ№м§•мңјлЎң н•ңлӢӨ. Thaker л“ұмқҳ м—°кө¬м—җ мқҳн•ҳл©ҙ нҠ№мҲҳ м ңмһ‘лҗң м•„нҒ¬лҰҙ мјҖмқҙмҠӨм—җ л§Өмқј 2мӢңк°„м”© к°җкёҲлҗң л§Ҳмҡ°мҠӨл“Өмқҳ мҠӨнҠёл ҲмҠӨ нҳёлҘҙлӘ¬мқё нҳҲмӨ‘ мҪ”лҘҙнӢ°мҪ”мҠӨн…ҢлЎ мқҖ мӢӨн—ҳ к°ңмӢң 3мқјл¶Җн„° мҰқк°Җн•ҳкё° мӢңмһ‘н•ҳм—¬ 7мқјкіј 14мқјм—җлҠ” лҢҖмЎ°кө°м—җ 비көҗн•ҳм—¬ мң мқҳн•ҳкІҢ мҰқк°Җн•ҳмҳҖлӢӨ. к·ёлҹ¬лӮҳ, мһҘкё°к°„мқҳ мҠӨнҠёл ҲмҠӨ м Ғмҡ©мқҙ нҳҲмӨ‘ мҪ”лҘҙнӢ°мҪ”мҠӨн…ҢлЎ мқ„ м§ҖмҶҚм ҒмңјлЎң лҶ’мқҖ мғҒнғңлЎң мң м§Җн•ҳм§ҖлҠ” м•ҠлҠ” кІғмңјлЎң ліҙмқёлӢӨ[
10]. Voorheesмқҳ м—°кө¬м—җ л”°лҘҙл©ҙ, н•ҳлЈЁ 6мӢңк°„м”© л¶ҖлҸҷ мҠӨнҠёл ҲмҠӨлҘј м Ғмҡ©н•ҳмҳҖмқ„ л•Ң м Ғмҡ© мҙҲкё°мқё 7мқјм—җ к°ҖмһҘ мҲҳм№ҳк°Җ лҶ’м•ҳмңјлӮҳ мқҙнӣ„ м җм°Ё к°җмҶҢн•ҳлҠ” 추세лҘј ліҙмҳҖлӢӨ[
26]. м§ҖмҶҚм Ғмқё мҠӨнҠёл ҲмҠӨк°Җ мһҘкё°м ҒмңјлЎң м Ғмҡ©лҗҳм—ҲмқҢм—җлҸ„ л¶Ҳкө¬н•ҳкі мҠӨнҠёл ҲмҠӨ нҳёлҘҙлӘ¬мқҙ к°җмҶҢн•ҳлҠ” мқҙмң мҷҖ кё°м „м—җ лҢҖн•ҙм„ңлҠ” м•Ңл Ө진 л°”к°Җ м—ҶмңјлӮҳ л‘җ к°Җм§Җмқҳ кІҪмҡ°лЎң м„ӨлӘ…мқҙ к°ҖлҠҘн• кІғмңјлЎң ліҙмқёлӢӨ. мІ«м§ё, м •мғҒм Ғмқё мҠӨнҠёл ҲмҠӨ нҳёлҘҙлӘ¬мқҖ лӮ®кіј л°Өмқҳ мқјмЈјкё°м—җ л”°лқј мҲҳм№ҳмқҳ кі м Җк°Җ мһҲлҠ” мқјліҖнҷ”(diurnal cycle)лҘј к°Җм§ҖлҠ” л°ҳл©ҙ, мһҘкё°м Ғ мҠӨнҠёл ҲмҠӨк°Җ м Ғмҡ©лҗЁм—җ л”°лқј мқјліҖнҷ”мқҳ мЈјкё°к°Җ лӢЁм¶• лҳҗлҠ” м§Җм—°мқҙ лҗ к°ҖлҠҘм„ұмқҙ мһҲлӢӨ. л‘ҳм§ё, мҠӨнҠёл ҲмҠӨ м Ғмҡ© мҙҲкё°м—җлҠ” мҰқк°Җн–ҲлҚҳ мҠӨнҠёл ҲмҠӨ нҳёлҘҙлӘ¬ мҲҳм№ҳк°Җ к°җкёҲмқ„ н•ҙм ңн•ҳлҠ” лҸҷмӢңм—җ кё°мӨҖм№ҳ(baseline)лЎң кёүкІ©нһҲ л–Ём–ҙм§ҖлҚҳ кІғмқҙ мҠӨнҠёл ҲмҠӨ м Ғмҡ©кё°к°„мқҙ кёём–ҙм§ҲмҲҳлЎқ 18мӢңк°„ мқҙмғҒ лҶ’мқҖ мҲҳм№ҳлҘј мң м§Җн•ҳлҠ” кІғмңјлЎң ліҙм•„[
26], л§Ңм„ұ мҠӨнҠёл ҲмҠӨлҠ” нҳёлҘҙлӘ¬мқҳ мҲҳм№ҳмқҳ мқјліҖнҷ”к°Җ м—Ҷм–ҙм§Җл©ҙм„ң мқјм • мҲҳмӨҖмқ„ м§ҖмҶҚм ҒмңјлЎң мң м§Җн• кІғмңјлЎң ліҙмқёлӢӨ. к·ёлҹ¬лӮҳ ліё м—°кө¬м—җм„ңлҠ” мҪ”лҘҙнӢ°мҪ”мҠӨн…ҢлЎ мқ„ мӢңк°„ к°„кІ©мқ„ л‘җкі мЈјкё°м ҒмңјлЎң мёЎм •н•ҳм§Җ м•Ҡм•ҳкі 8мЈјмҷҖ 16мЈјм—җ к°Ғк°Ғ нҡЎлӢЁм ҒмңјлЎң мёЎм •н•ҳмҳҖкё° л•Ңл¬ём—җ нҳёлҘҙлӘ¬мқҳ мқјліҖнҷ” мЈјкё°м—җ ліҖнҷ”к°Җ мһҲм—ҲлӢӨл©ҙ мқҙлҘј лӢЁл©ҙ 비көҗн•ҳкё°м—җ м ңн•ңмқҙ мһҲм—Ҳмқ„ кІғмңјлЎң 여겨진лӢӨ.
м—¬лҹ¬ мһ„мғҒм—°кө¬м—җм„ң мҠӨнҠёл ҲмҠӨлҠ” 비л§Ңмқҳ мӣҗмқё мӨ‘ н•ҳлӮҳмқё кІғмңјлЎң мЈјлӘ©лҗҳм–ҙмҷ”лӢӨ. мІҙлӮҙ мҪ”нӢ°мҶ”мқҳ кіјлӢӨ분비м—җ мқҳн•ң мҝ мӢұмҰқнӣ„кө° нҷҳмһҗмқҳ мҰқмғҒм—җм„ңмҷҖ л§Ҳм°¬к°Җм§ҖлЎң мҪ”нӢ°мҶ”мқҳ мҰқк°ҖлҠ” нҸ¬лҸ„лӢ№мӢ н•©м„ұ(gluconeogenesis)мҷҖ м§Җм§Ҳ분н•ҙлҘј мң лҸ„н•ҳкі , л ҷнӢҙ м Җн•ӯм„ұмқ„ мҰқк°ҖмӢңмјң мІҙмӨ‘мқҳ мҰқк°ҖмҷҖ кі м—ҙлҹү мқҢмӢқм—җ лҚ” нғҗлӢүн•ҳкІҢ н•ҳлҠ” н–үлҸҷ ліҖнҷ”лҘј мқјмңјнӮЁлӢӨ[
27]. к·ёлҹ¬лӮҳ мӮ¬лһҢмқҳ кІҪмҡ°мҷҖлҠ” лӢӨлҘҙкІҢ ліё м—°кө¬ кІ°кіјлҠ” мҠӨнҠёл ҲмҠӨ м Ғмҡ©мқҙ л ҷнӢҙ мҲҳм№ҳк°Җ мҰқк°Җн•ҳкі мӢқмқҙнҡЁмңЁмқҙ л–Ём–ҙмЎҢмқҢм—җлҸ„ л¶Ҳкө¬н•ҳкі мІҙмӨ‘кіј нҳҲмӨ‘ м§Җн‘ңл“Өмқҙ к°җмҶҢн•ҳлҠ” кІғмңјлЎң лӮҳнғҖлӮҳ мҠӨнҠёл ҲмҠӨмқҳ м Ғмҡ© л°©лІ•мқҙлӮҳ кё°к°„мқҙ м Ғн•©н•ҳм§Җ м•Ҡм•ҳмқ„ к°ҖлҠҘм„ұмқ„ ліҙм—¬мЈјкі мһҲлӢӨ. мқҙлҠ” м§Җл°© мЎ°м§Ғкіј к°„мқҳ лҢҖмӮ¬ кҙҖл Ё мң м „мһҗл“Өмқҳ мң мқҳн•ң ліҖнҷ”к°Җ ліҙмқҙм§Җ м•Ҡкі мҳӨнһҲл Ө м§Җл°©мӮ°мқҳ лІ нғҖ мӮ°нҷ”лҘј мҙү진н•ҳлҠ” кІ°кіјмҷҖ мқјм№ҳн•ҳкі мһҲлӢӨ. м•„мҡёлҹ¬ мҠӨнҠёл ҲмҠӨлҘј м Ғмҡ©н•ң мғқмҘҗл“Өмқҳ мғқмІҙмӢңкі„ мң м „мһҗ л°ңнҳ„мқҙ 45% кі м§Җл°©мӢқмқҙлҘј мӨҖ мғқмҘҗл“Өмқҳ мҲҳм№ҳм—җ 비н•ҳм—¬ к°җмҶҢн•ң кІғмқҖ м§Җл°©н•©м„ұмқҙ мӨ„кі мҶҢлӘЁк°Җ лҠҳм–ҙлӮЁм—җ л”°лқј мғқмІҙм§Җн‘ңл“Өмқҙ мҳӨнһҲл Ө к°ңм„ лҗҳлҠ” кІғкіј м—°кҙҖм„ұмқҙ мһҲлҠ” кІғмңјлЎң ліҙмқёлӢӨ.
лӢ№лҢҖмӮ¬мҷҖ м§Җл°©лҢҖмӮ¬к°Җ л§җмҙҲ мғқмІҙмӢңкі„ мң м „мһҗмқҳ мЎ°м Ҳм—җ м–ҙл–Ө кё°м „мңјлЎң мһ‘мҡ©н•ҳлҠ”м§ҖлҠ” м•„м§Ғк№Ңм§Җ мһҳ м•Ңл Өм ёмһҲм§Җ м•ҠлӢӨ. л¬ҙм—ҮліҙлӢӨ лҢҖмӮ¬м—җ кҙҖм—¬н•ҳлҠ” мһҘкё°л“Өм—җ лӢЁмқј м„ёнҸ¬мҲҳмӨҖмқҳ л§җмҙҲ мғқмІҙмӢңкі„ мң м „мһҗл“Өмқҙ мЎҙмһ¬н•ҳл©° мҲҳл©ҙ-к°Ғм„ұ мЈјкё°мҷҖлҠ” лі„к°ңлЎң мЎ°м§Ғ л°Ҹ кё°кҙҖ лі„ нҠ№мқҙм Ғмқё мғқмІҙлҰ¬л“¬мқ„ мғқм„ұн• кІғмқҙлқјлҠ” к°ңл…җмқҙ мөңк·јм—җ лҢҖл‘җн•ҳмҳҖкё° л•Ңл¬ём—җ нҳ„мһ¬к№Ңм§ҖлҠ” мғқмІҙмӢңкі„ мң м „мһҗ мЎ°мһ‘мқ„ мқҙмҡ©н•ҳкұ°лӮҳ мҲҳл©ҙ-к°Ғм„ұ мЈјкё°лҘј көҗлһҖмӢңнӮЁ нӣ„ мғқлҰ¬м Ғ ліҖнҷ”лҘј кҙҖм°°н•ҳлҠ” м—°кө¬л“Өмқҙ лҢҖл¶Җ분мқ„ м°Ём§Җн•ҳкі мһҲлӢӨ. мқҙлҘј л’·л°ӣм№Ён•ҳлҠ” кё°м „мқҖ мғқмІҙмӢңкі„лҘј мЎ°м Ҳн•ҳлҠ”
CLOCK,
BMAL1мқҙ м „мӮ¬мқёмһҗ(transcription factor)мқҙл©°
CRY,
PER л“ұмқҙ мЎ°м Ҳмқ„ л°ӣлҠ” н•ҳмң„ мң м „мһҗл“ӨмқҙлҜҖлЎң м§Җл°©мғқм„ұм—җ кҙҖм—¬н•ҳлҠ” sterol regulatory element-binding protein (SREBP) лӮҳ CCAAT-enhancer-binding proteins (
C/EBPs) л“ұмқҙ н‘ңм Ғмң м „мһҗлЎңм„ң мғқмІҙмӢңкі„мқҳ мЎ°м Ҳмқ„ л°ӣлҠ” кІғмңјлЎң м„ӨлӘ…н•ҳкі мһҲлӢӨ[
28]. к·ёлҹ¬лӮҳ ліё м—°кө¬мІҳлҹј кі м§Җл°©мӢқмқҙ л“ұмқ„ нҶөн•ҙ 비л§Ңмқ„ мң лҸ„н•ҳмҳҖмқ„ л•Ңм—җлҸ„ мғқмІҙмӢңкі„ мң м „мһҗмқҳ ліҖнҷ”к°Җ мқјм–ҙлӮҳлҠ” м—°кө¬кІ°кіјлҸ„ мһҲм–ҙ[
29] мғҒнҳём Ғмқё мЎ°м Ҳмқҙ мқјм–ҙлӮҳлҠ” кІғмңјлЎң ліҙмқёлӢӨ. м•„м§Ғк№Ңм§Җ л§җмҙҲ мғқмІҙмӢңкі„ мң м „мһҗмқҳ мғҒмң„ мӢ нҳём „лӢ¬мІҙкі„к°Җ нҷ•мӢӨнһҲ м•Ңл Ө진 кІғмқҙ м—ҶмңјлӮҳ мқјл¶Җ м—°кө¬м—җм„ң м—җл„Ҳм§Җ н•ӯмғҒм„ұмқ„ мЎ°м Ҳн•ҳлҠ” AMP-activated protein kinase (AMPK)мқҳ нҷңм„ұмқҙ
PPARs кіј к°ҷмқҙ лҢҖмӮ¬м—җ кҙҖм—¬н•ҳлҠ” мЈјмҡ” м „мӮ¬мқёмһҗмқҳ нҷңм„ұмқ„ мң лҸ„н•ҳкі мқҙл“Өмқҳ н‘ңм Ғ мң м „мһҗлЎң мғқмІҙмӢңкі„ мң м „мһҗл“Өмқҳ л°ңнҳ„мқҙ мқјм–ҙлӮҳлҠ” кІғмңјлЎң ліҙкі лҗҳкі мһҲлӢӨ[
6]. к·ёлҹ¬лӮҳ мқҙлҘј м§Җм§Җн•ҳлҠ” м—°кө¬л“Өмқҙ мҶҢмҲҳмқҙлҜҖлЎң м •нҷ•н•ң кё°м „мқ„ к·ңлӘ…н•ҳкё° мң„н•ң 추к°Җм Ғмқё м—°кө¬к°Җ н•„мҡ”н• кІғмқҙлӢӨ.



 PDF Links
PDF Links PubReader
PubReader Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print